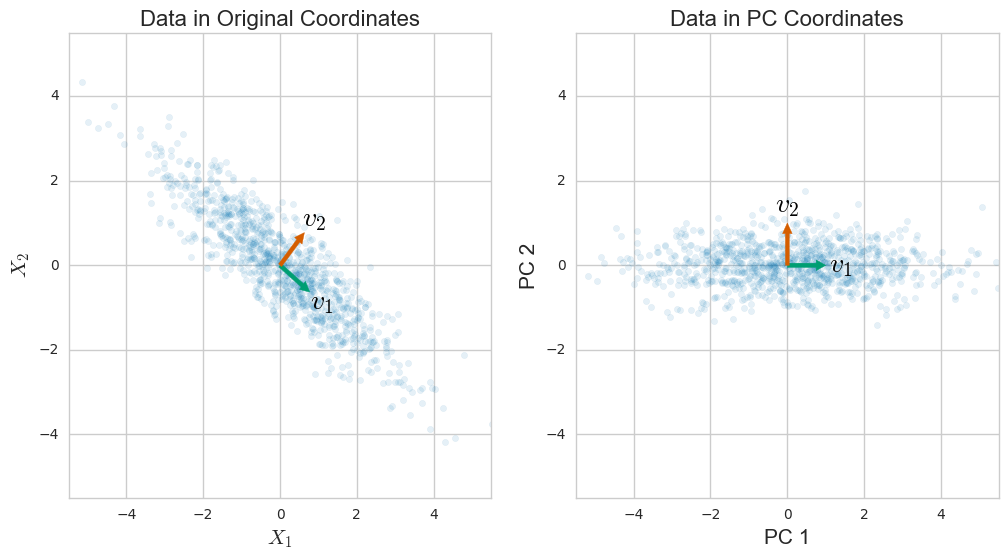
Нехай матриця даних має розмір n × p , де n - кількість вибірок, а p - кількість змінних. Припустимо, що він по центру , тобто засоби стовпця були відняті і тепер дорівнюють нулю.Хn × pнp
Тоді ковариационной матриці C задається C = Х ⊤ Х / ( п - 1 ) . Це симетрична матриця, і тому її можна діагоналізувати: C = V L V ⊤ , де V - матриця власних векторів (кожен стовпець - власний вектор), а L - діагональна матриця з власними значеннями λ i у порядку зменшення по діагоналі . Власні вектори називають головними осями або основними напрямкамиp × pСC = X⊤X / (n-1)
C = V L V⊤,
VLλiданих. Проекції даних на головні осі називаються
основними компонентами , також відомими як
оцінки ПК ; їх можна розглядати як нові, перетворені, змінні.
-го основного компонента задається
J -го стовпця
X V . Координати
я -й точка даних в новому просторі ПК задаються
я -й рядком
X V .
jjX ViiX V
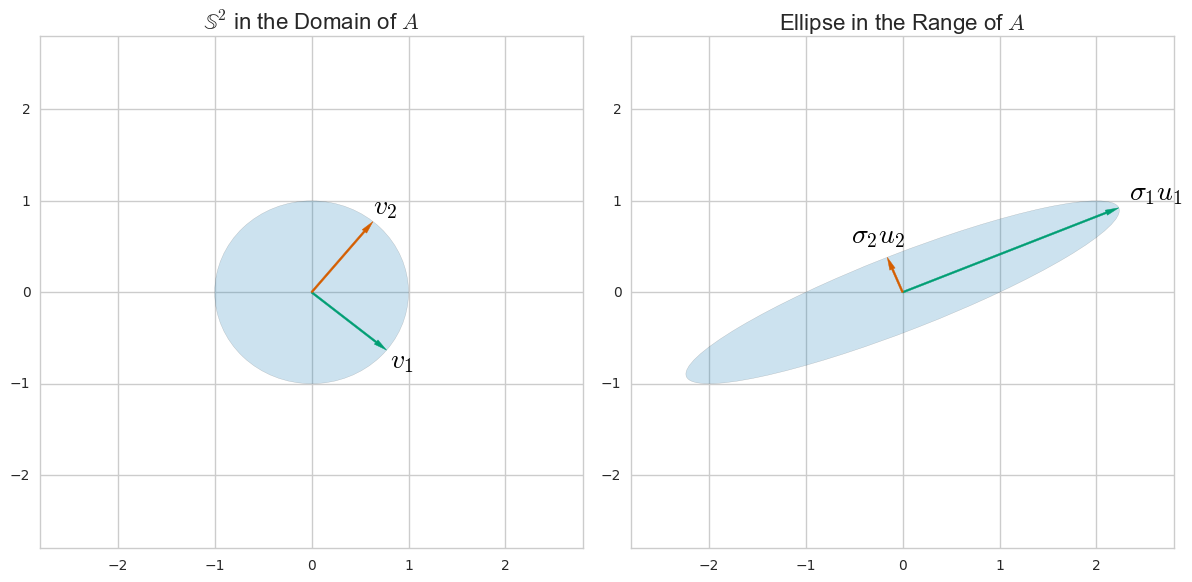
Якщо ми зараз проведемо сингулярне розкладання значення , отримаємо розклад X = U S V ⊤ , де U - унітарна матриця, а S - діагональна матриця сингулярних значень s i . Звідси легко видно, що C = V S U ⊤ U S V ⊤ / ( n - 1 ) = V S 2Х
X = U S V⊤,
USсiозначає, що правильні сингулярні вектори
Vє основними напрямками і що сингулярні значення пов'язані з власними значеннями матриці коваріації через
λi=s 2 i /(n-1). Основні компоненти задаються
XV=USV⊤V=US.
C = V S U⊤U S V⊤/ (n-1)= V S2n - 1V⊤,
Vλi= s2i/ (n-1)X V = U S V⊤V = U S
Узагальнити:
- Якщо , то стовпці V - основні напрямки / осі.X = U S V⊤V
- Стовпці це основні компоненти ("бали").U S
- Сингулярні значення пов'язані з власними значеннями матриці коваріації через . Власні значення λ я показую дисперсії відповідних ПК.λi= s2i/ (n-1)λi
- Стандартизовані бали подаються стовпцями і навантаження задаються стовпцямиVS/ √n - 1-----√U . Дивіться, наприклад,тутітут,чому "навантаження" не слід плутати з основними напрямками.V S / n - 1-----√
- Сказане вище правильне, лише якщо по центру розміщено ХТільки тоді матриця коваріації дорівнює .Х⊤X / (n-1)
- Сказане вище правильне лише для мають зразки у рядках та змінні у стовпцях. Якщо змінні знаходяться у рядках, а зразки - у стовпцях, то U та V обмінюються інтерпретаціями.ХUV
- Якщо потрібно виконати PCA на кореляційній матриці (замість коваріаційної матриці), то стовпці повинні бути не лише центрировані, а й стандартизовані, тобто поділені на їх стандартні відхилення.Х
- Щоб зменшити розмірність даних від до K < р , виберіть K перші стовпчики U і K × K верхня ліва частина S . Їх добуток U k S k є необхідною матрицею n × k, що містить перші k ПК.pк < ркUk × kSUkSkn×kk
- kV⊤kXk=U⊤kS⊤kV⊤kn×pkXkk
- Un×nVp×pn>pn−pUSUn×pn≫pUn≪p
Подальші посилання